IPCW的SAS实现
IPCW
前面我们已经在这篇文章里介绍过IPCW的原理。当时有小伙伴在知识星球里提问,IPCW在sas中的具体实现。
今天我们来大致讲一下它的SAScode,完整的代码已经上传进我的星球,感兴趣的同学可以自行下载查看。
数据结构
首先,我们需要准备count格式的生存数据,如下面这个dummy dataset:
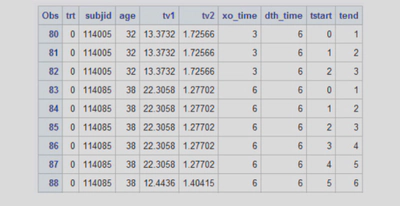
其中,xo_time标示crossover发生的时间,即表示是否发生了crossover,tstart、tstop标明count格式的生存数据,显示各个时间点受试者at risk的情况。
step 1: 计算未删失(未发生crossover)的概率
这一步,根据协变量的不同,采用的模型也不行同。对于包含时依协变量的数据,我们可以分别构建包含基线协变量以及包含时依协变量的两个模型,来计算权重。
采用logistic可以使用以下code:
proc genmod data=dummy descending order=data;
class crossover id;
model crossover= baseline_covariates time_dependent_covariates / d=bin link=logit;
repeated subject=id;
output out=out prob=t;
run;
我们在星球里给的代码采用的是cox来计算概率,如下:
proc phreg data=weight_est outest=survmod covs(aggregate) covm; * covs option and id statement adjust for repeated measures;
by &trt;
class &bl_cl &tv_cl;
model (&tstart,&tend)*xo_cnsr(0)= &bl_ct &bl_cl &tv_ct &tv_cl /rl;
id &subjid;
output out= probs_TV survival = w_TV;
run;
step2: 利用IPCW加权
需要注意的是,在加权之前,我们可以进行权重的截断来进行stablize。
proc phreg data=surv_ph outest=survmod covs(aggregate) covm; * covs option and id statement adjust for repeated measures;
title 'Final Model using weights from Cox PH Model';
class &bl_cl &tv_cl &trt(ref=first);
model (&tstart,&tend)*dth_event(0)= &trt &bl_ct &bl_cl &tv_cl &tv_ct /rl;
id &subjid;
weight sw_t/norm;
run;