MMRM的SAS和R实现
missing data
在星球里,不断有小伙伴提问到missing data相关的问题。

missing data是个庞大的问题,我们只能在遇到具体的问题时对应给出回答,再争取由点到面。
一般,具体的思路如下:
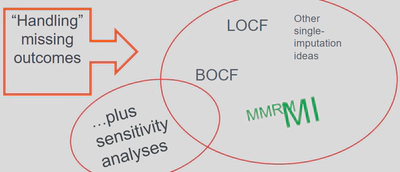
今天我们主要讲一讲MMRM(mixed model repeated measures)。
MMRM
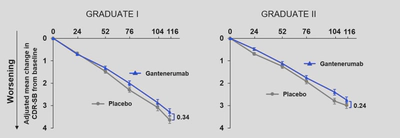
对于logitudinal data和重复测量数据,我们可以采用MMRM来分析。MMRM的基本思路是,将每个受试者的测量数据建模,然后将所有受试者的模型汇总,得到整体的模型。MMRM不具体地指定个体水平上的random effects,相反,其基于时间刻度上重复测量之间的相关性(残差相关)进行建模(不仅是估计means同时估计variances和covariances)。对于这个相关性的刻画,MMRM有着灵活的相关矩阵结构的假设,如unstructured (UN)、homogeneous Toeplitz (TOEP)、auto-regressive (AR(1))、heterogeneous auto-regressive (ARH(1))、compound symmetry (CS)、heterogeneous compound symmetry (CSH)等,可以用来估计固定效应,一般我们选择UN。而对于固定效应,MMRM可以估计每组每个时间节点endpoint的均值水平,如来自NEJM的下图。
而更为重要的是,MMRM可以基于所有现有的数据,包括missing data(missing at random),来估计固定效应。这一点是MMRM的优势,也是我们选择MMRM的原因。
下面我们将给出MMRM的SAS和R的实现。这里,我们dummy一个RCT的数据,两组,每组200个受试者,每个受试者有9周的测量数据(分值),无缺失,需要比较两组第9周分值相对于baseline改变(change from baseline)之间的差异,采用MMRM分析。
SAS
proc mixed data=adqs method=reml covtest;
class usubjid avisit trt01p(REF='Placebo') region1;
model chg = trt01p avisit base region1 trt01p*avisit / ddfm=KR;
repeated avisit / subject=usubjid type=un;
lsmeans trt01p * avisit / cl pdiff alpha = 0.05 obsmargins;
run;
除模型诊断等相关信息之外,主要least suqares means结果如下:

R
library(mmrm)
library(emmeans)
library(dplyr)
fit_mmrm <- mmrm(
formula = CHG ~ TRT01P + AVISIT + BASE + REGION1 + TRT01P*AVISIT +
us(AVISIT | USUBJID),
data = adqs,
reml = TRUE,
control = mmrm_control(method = "Kenward-Roger" , vcov ="Kenward-RogerLinear")
)
summary(fit_mmrm)
二者结果基本一致,需要注意的是,在R中,利用以上代码的话,如果要获得组间LSM difference,我们需要另外编程来获得,具体的代码已经放进星球,欢迎大家下载查阅。